Les Programmes de recherches
Le génome humain code pour plus de 500 protéines kinases (ensemble appelé « kinome ») qui interviennent dans la régulation d’une multitude de voies cellulaires. L’inhibition de ces enzymes clées est devenue, l’un des premiers choix dans le développement de stratégies thérapeutiques ciblées pour les maladies néoplasiques. A présent, 8% du kinome a été ciblé, conduisant au développement de nouveaux principes actifs médicamenteux. Néanmoins, les travaux d’investigation visant à identifier ou caractériser des cibles kinases pour des applications thérapeutiques sont très hétérogènes.
Ainsi, une catégorie de kinase appelée « dark » kinases a été créée afin de regrouper des cibles pour lesquelles peu de travaux de recherche ont été réalisés. A ce jour, aucune relation entre ces kinases et une maladie n’a encore été mise en évidence.
Toutefois des études bio-informatiques récentes révèlent que cette catégorie présente un haut potentiel dans la découverte de nouvelles cibles applicables en thérapie anticancéreuse. Notre équipe souhaite pouvoir réaliser une exploration, de cette catégorie de kinases sous-étudiées, par des techniques prédictives de recherche de substrat phosphorylable et d’évaluer leurs implications dans des voies de régulation cellulaire.
Nous proposons également par cette méthode de mettre en place une stratégie de criblage moléculaire visant l’identification d’inhibiteurs pharmacologiques spécifique
Ce projet est financé par la Ligue Contre le Cancer et piloté par Thomas Robert et Morgan Lelièvre
Projet ANR AtropCN-DRUGS (2020-2024)
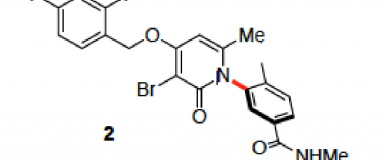
Après plusieurs années de travail collaboratif avec l’équipe du professeur Francoise Colobert (ECPM/Université de Strasbourg), Nous avons eu l’honneur d’obtenir le financement par l’ANR pour le projet AtropCN-drug (2020-2024)
L'idée de ce projet repose sur un défi en synthèse chimique : la possibilité de produire des molécules structurées dans l'espace avec une chiralité selon un axe C-N (exemple de molécule attachée). Peu d'équipe dans le monde sont capables de réaliser des synthèses énantiosélectives de cette classe de produits. Ce domaine est d'un grand intérêt en chimie médicinale en fournissant aux biologistes des structures complètement originales aux propriétés encore inexplorées.
La plateforme de criblage KISSf est partenaire de ce projet car certaines kinases se sont avérées sensibles à des produits pionniers de synthèse.
Projet Cathalymar (2018-2020) cloturé
La plateforme KISSf s’est impliquée dans le projet CatalyMar. C’est un projet interrégional Bretagne et Pays de la Loire qui a pour objectif d’associer 9 plateformes impliquées dans plusieurs axes du réseau Biogenouest, autour d’une source de biomolécules innovantes : les micromycètes marins. L’objectif est de créer un réseau de collaboration entre plusieurs plateformes afin de développer un pipeline de bioprospection et de valorisation d’espèces marines abondantes et inexploitées. Le projet a débuté en novembre 2019, la durée du projet est de 2 ans. En juillet 2020 la plateforme KISSf a reçu de la plateforme Thalassomics (Nantes) 48 extraits bruts. Ceux-ci ont été analysés en première intention à 2 concentrations (5 et 50 µg/mL) sur un panel de 8 kinases représentatif du Kinôme pour une recherche d’activité inhibitrice de protéines kinases. Des activités ont été détectées dans certains extraits, pour la majorité d’entre eux issus de Penicillium. Les 48 extraits ont également été analysés par BIODIMAR pour l’identification d’activités anti-oxydantes, anti-microbiennes et anti-fouling. Une compilation des différents résultats obtenus va permettre de sélectionner 5 à 10 extraits qui seront fractionnés pour identifier les principes actifs des extraits retenus.
Stéphane Bach et Blandine Baratte sont les interlocuteurs de KISSf pour ce projet.